Risultati
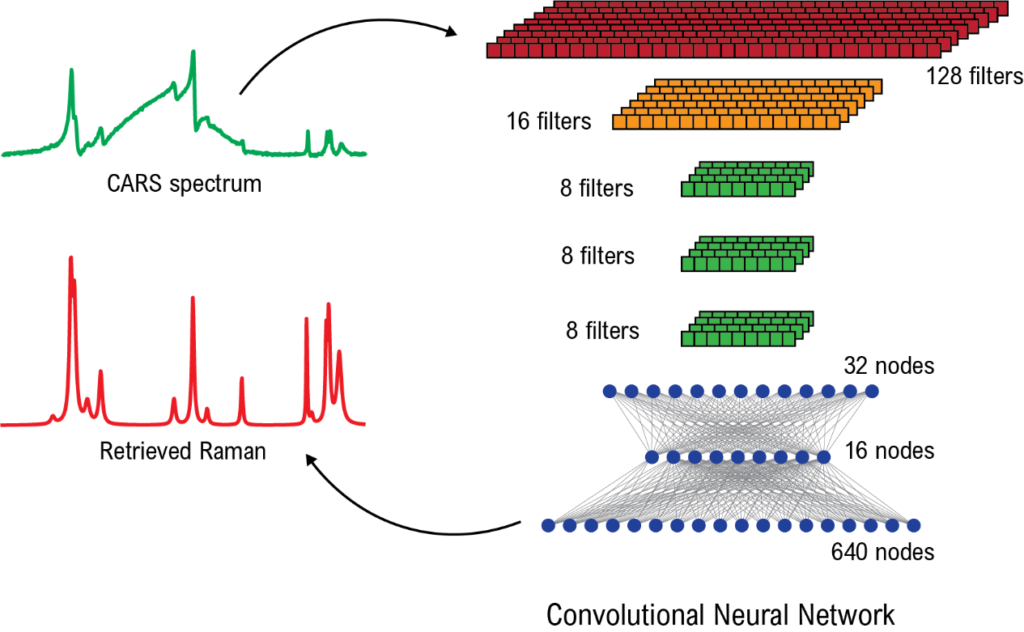
Nell’ambito della microscopia vibrazionale non-lineare, ogni pixel di un’immagine consta di una serie di valori, collettivamente chiamati spettro. I valori dello spettro sono correlati con la concentrazione di specie chimiche nella porzione di spazio identificata dal pixel. Uno dei problemi nell’ambito della microscopia vibrazionale CARS (Coherent Anti-Stokes Raman Scattering), che tra le complesse tecniche non-lineari è una delle più promettenti, è la presenza di un segnale spurio, che si somma a quello fisicamente rilevante, e distorce e degrada lo spettro, impedendo una corretta quantificazione e identificazione dei legami chimici.
Nel lavoro di Valensise, Giuseppi, Vernuccio, De la Cadena, Cerullo e Polli, “Removing non-resonant background from CARS spectra via deep learning”, pubblicato sulla prestigiosa rivista APL photonics (https://doi.org/10.1063/5.0007821) il deep learning, una delle tecniche più impiegate di intelligenza artificiale, è impiegato per rimuovere il contributo spurio dagli spettri CARS, misurati in laboratorio al Politecnico di Milano, sfruttando un setup interamente sviluppato dal gruppo di ricerca del Prof. Polli. In figura è rappresentato il concetto dell’articolo. Lo spettro misurato viene processato da una rete neurale convoluzionaria opportunamente allenata a restituire lo spettro pulito dai segnali spuri.
Rispetto alle tecniche tradizionali, il filtraggio del rumore basato sul deep learning promette tempi di processamento più veloci e maggiore versatilità, oltre ad una potenziale applicazione in tempo reale, per ricostruire immagini chimiche quantitativamente affidabili immediatamente dopo che esse siano acquisite.
Questo risultato, ottenuto grazie ai finanziamenti del progetto NEWMED, costituisce un importante milestone per il raggiungimento dell’obiettivo 1: “Sviluppo di microscopi Raman per istopatologia intraoperatoria di lesioni tumorali”

L’osservazione in tempo reale ed in modalità non invasiva di campioni biologici vitali è una delle tematiche centrali della microscopia. La microscopia multi-fotone permette di studiare i meccanismi alla base di diverse funzioni cellulari, combinando diverse modalità tra loro come, la fluorescenza a due fotoni (TPEF), la generazione di seconda e terza armonica (SHG-THG) e la microscopia Raman coerente (CARS ed SRS), in assenza di marcatori esogeni. In un lavoro recentemente pubblicato sulla rivista internazionale “Frontiers in Bioengineering and Biotechnology” (V. Parodi, E. Jacchetti, R. Osellame, G. Cerullo, D. Polli and M.T. Raimondi, “Nonlinear Optical Microscopy: From Fundamentals to Applications in Live Bioimaging”, Front. Bioeng. Biotechnol. 8, Article 585363 (2020)) abbiamo raccolto esempi recenti dell’utilizzo di queste tecniche osservazionali per studiare ad esempio la proliferazione tumorale, il differenziamento cellulare e la rigenerazione di un tessuto in tessuti popolati da singole specie cellulari in vitro ed in tessuti eterogenei direttamente nell’animale nel loro stato imperturbato ed in vitalità. Infine, abbiamo dimostrato che la microscopia CARS e SHG è una valida alternativa alle tecniche istopatologiche per la caratterizzazione del differenziamento di cellule staminali in adipogenesi e condrogenesi in un ambiente 3D ingegnerizzato in vitro.
Questo risultato, ottenuto grazie ai finanziamenti del progetto NEWMED, costituisce un importante milestone per il raggiungimento dell’obiettivo 1: “Sviluppo di microscopi Raman per istopatologia intraoperatoria di lesioni tumorali”

Recenti studi in vitro hanno dimostrato che la scelta della topografia e del design 3D del substrato di coltura è in grado di determinare il destino cellulare, ovvero l’espressione del fenotipo delle cellule staminali ad esso adese, attraverso specifici stimoli meccanici. Tuttavia, i meccanismi alla base della traduzione degli sforzi in segnali biochimici non sono ancora chiari. Nel lavoro di Parodi e colleghi “Characterization of mesenchymal stem cell differentiation within miniaturized 3D scaffolds through advanced microscopy techniques”, pubblicato sulla rivista International Journal of Molecular Sciences 21, 8498, 2020, abbiamo utilizzato un substrato tridimensionale, il Nichoid, per valutarne l’effetto durante il differenziamento di cellule staminali mesenchimali verso il fenotipo adipogenico e condrogenico. In questo lavoro abbiamo confrontato la microscopia confocale a fluorescenza con le tecniche nonlineari CARS ed SHG ed abbiamo dimostrato che il Nichoid stimola la produzione di grandi vescicole lipidiche ben visibili attraverso CARS e limita la condrogenesi in quanto una grande quantità di collagene è visibile esternamente alla microstruttura con SHG.
Questo risultato, ottenuto grazie ai finanziamenti del progetto NEWMED, costituisce un importante milestone per il raggiungimento dell’obiettivo 1: “Sviluppo di microscopi Raman per istopatologia intraoperatoria di lesioni tumorali”
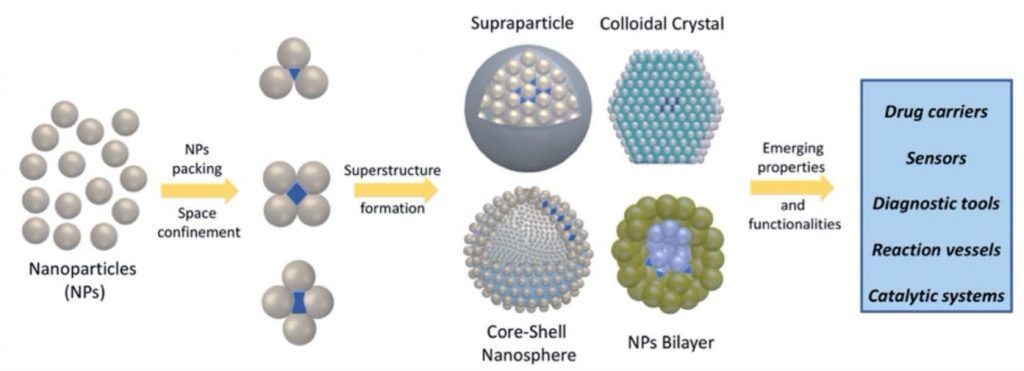
L’auto-assemblaggio di nanoparticelle (NP) ha portato allo sviluppo di un insieme di sistemi funzionali di natura nanomerica con diverse architetture e funzioni. Il lavoro di Metrangolo, Baldelli Bombelli, Pigliacelli, Dichiarante, pubblicato sulla prestigiosa rivista “Chemical Science” (https://pubs.rsc.org/en/content/articlehtml/2021/sc/d0sc05697a), discute il disegno e l’applicazione di soprastrutture di nanoparticelle (SPs), caratterizzate da compartimenti nanoconfinati formati in seguito al meccanismo di auto-assemblaggio e riporta una panoramica delle strategie di sintesi delle SP ad oggi conosciute e del ruolo delle nanocavità confinate in applicazioni in diversi campi. Inoltre, gli autori forniscono un contributo rispetto allo sviluppo di SP con nano compartimentazioni più avanzate, in grado di agire in maniera dinamica. Le opportunità di avanzamenti futuri nell’ambito dell’auto-assemblaggio di NP e di SP sono ampiamente discusse nell’articolo. Gli autori ringraziano il progetto NEWMED per i finanziamenti ricevuti. Questa review pone le basi per ulteriori future ricerche riguardo all’utilizzo di SP come sistemi per il rilascio controllato di farmaci, in linea con l’obiettivo 3 del progetto NEWMED.
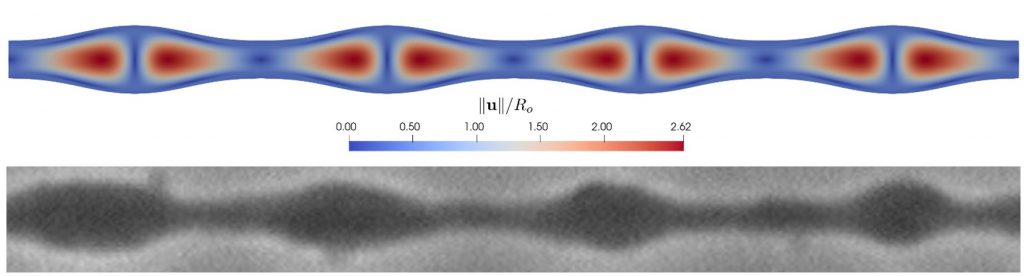
Diverse patologie, dalla malattia di Alzheimer alle infezioni da coronavirus, causano delle modifiche nella struttura dei neuroni portando alla formazione di rigonfiamenti periodici lungo gli assoni. Evidenze sperimentali mostrano che tali cambiamenti morfologici sono correlati a una drastica riduzione dei microtubuli presenti nell’assoplasma. In questo lavoro viene proposto un modello matematico di tale fenomeno basato sulla meccanica dei continui. L’assone viene modellizzato come un cilindro elastico circondato da una guaina, la corteccia di actina, capace di contrarsi attivamente.
Si dimostra che, al diminuire della rigidezza elastica dell’assoplasma, l’assone va incontro ad un’instabilità meccanica che conduce alla formazione di rigonfiamenti periodici. Tale instabilità è causata dalla contrattilità della corteccia di actina e dalla depolimerizzazione della rete di microtubuli dell’assoplasma. I risultati di questa ricerca contribuiscono alla comprensione degli effetti sui neuroni di svariate patologie, neurodegenerative e virali, e possono essere di aiuto nella ricerca di nuove strategie di cura e trattamento.
Il risultato è stato pubblicato sulla rivista Physical Review E.
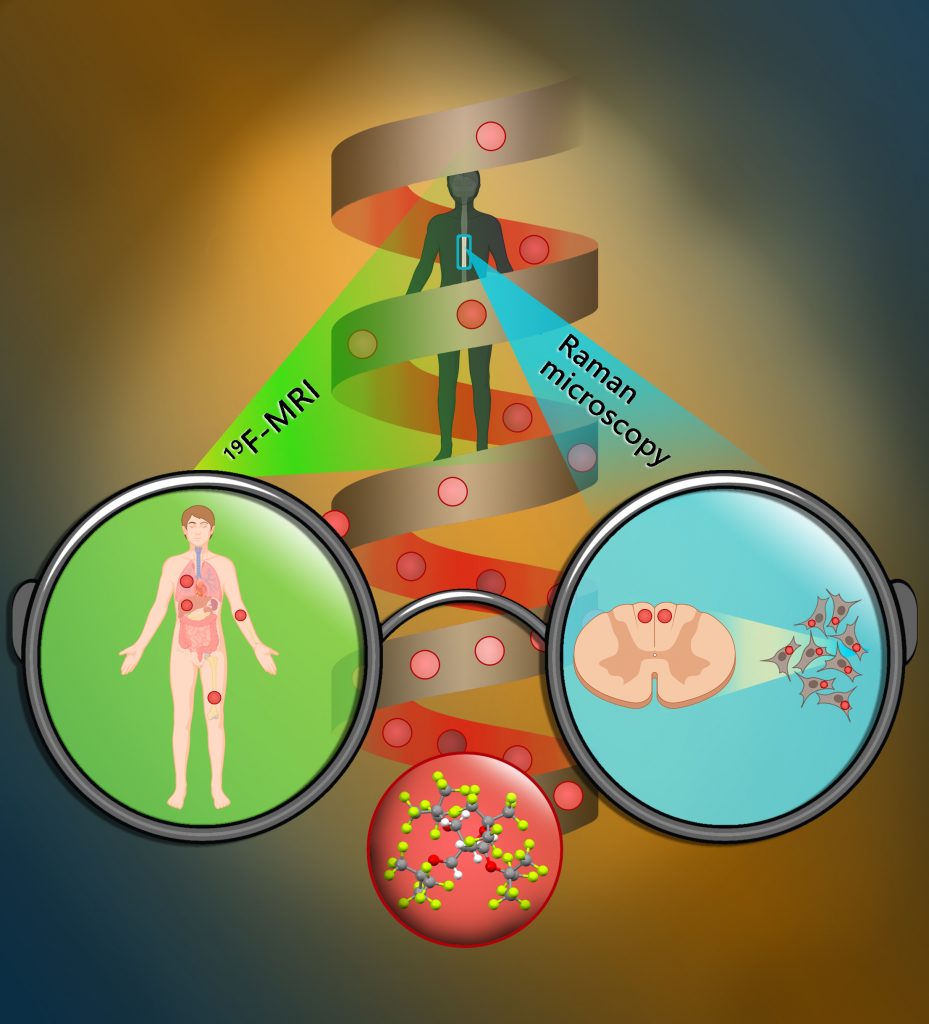
Nano-sonde per permettere diagnosi più efficaci
Uno studio condotto da Politecnico di Milano, Consiglio Nazionale delle Ricerche, IRCCS Istituti Clinici Scientifici Maugeri e IRCCS Ospedale San Raffaele è stato pubblicato sulla prestigiosa rivista «Journal of the American Chemical Society» (JACS); i ricercatori hanno sviluppato un nuovo tipo di nano-sonde fluorurate che permettono di visualizzare a diversi livelli di risoluzione l’attività cellulare, grazie alla combinazione di imaging di risonanza magnetica e microscopia Raman.
Avere a disposizione un’unica sonda in grado di essere registrata sia in vivo attraverso la risonanza magnetica che, con maggiore dettaglio, nei tessuti e all’interno di cellule target tramite microscopia Raman potrebbe offrire a breve la possibilità di seguire in dettaglio il percorso di un determinato farmaco dal sito di somministrazione fino alla singola cellula.
La ricerca, che ha visto la collaborazione dei ricercatori del Dipartimento di Chimica, Materiali e Ingegneria Chimica “Giulio Natta” e de Dipartimento di Fisica del Politecnico nasce nel contesto nel progetto NEWMED, finanziato da Regione Lombardia, che ha come obbiettivo l’introduzione di nuovi approcci di nanomedicina e di fotonica nel contesto clinico.
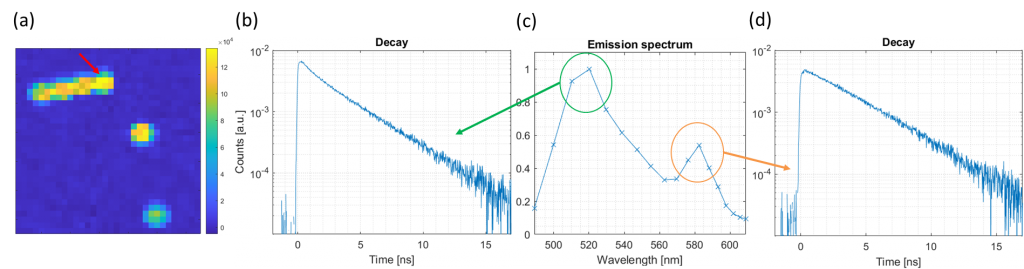
Molte molecole di interesse biologico (e.g. il NADH) emettono fluorescenza a seguito di un’eccitazione con un impulso di luce. Lo studio della fluorescenza fornisce informazioni sui processi fotofisici a livello molecolare che avvengono ad esempio in una cellula. In particolare, lo spettro di emissione di fluorescenza, il tempo di vita e la localizzazione della molecola emettitrice costituiscono un insieme di parametri ad alto contenuto informativo. In un lavoro pubblicato recentemente sulla rivista internazionale Optics Letters (A. Ghezzi, A. Farina, A. Bassi, G. Valentini, I. Labanca, G. Acconcia, I. Rech, C. D’Andrea “Multispectral compressive fluorescence lifetime imaging microscopy with a SPAD array detector”) abbiamo realizzato un nuovo tipo di microscopio, in grado di acquisire contemporaneamente i parametri spaziali, temporali e spettrali del campo di vista. L’acquisizione avviene in modo efficace, grazie ad una strategia di misura che consente di ridurre il numero di acquisizioni necessarie a pari contenuto informativo del dato. Uno degli elementi chiave del lavoro è un nuovo tipo di rivelatore di fotoni, realizzato dal Dipartimento di Elettronica, grazie alla cui architettura è stato possibile aumentare l’efficienza di conteggio dei singoli fotoni rispetto all’attuale stato dell’arte tecnologico. Per dimostrare la tecnica, abbiamo raccolto dati di fluorescenza da un vetrino da microscopia contente microanelli emettitori.
L’architettura del microscopio è fondamentale per studiare i processi biologici e permette di sviluppare metodologie di acquisizione di immagini facilmente scalabili ai sistemi macro. Questo risultato, ottenuto grazie ai finanziamenti del progetto NEWMED, costituisce un importante milestone nello studio e nell’implementazione delle tecniche di compressive sensing, che costituiscono un elemento centrale nel progetto. L’acquisizione di immagini funzionali che, oltre alle dimensioni spaziali X,Y, contengono informazioni spettrali e temporali, introduce un paradigma innovativo nello studio dei processi biologici, dalla microscala alla scala dei tessuti e degli organi
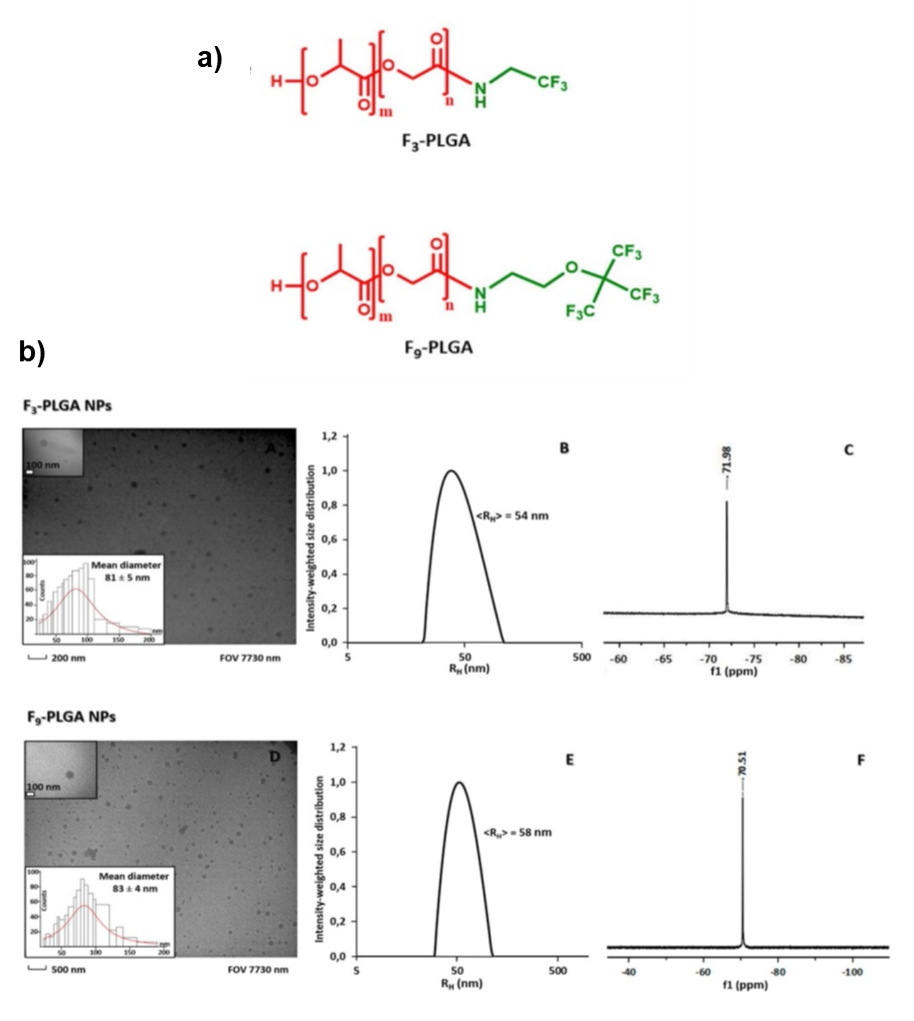
Nella continua ricerca di sviluppare sistemi in grado di contenere allo stesso tempo funzioni diagnostiche e terapeutiche, numeri sforzi sono stati rivolti alla progettazione di sistemi ‘multifunzionali’ per il rilascio di farmaci nel trattamento di diverse patologie. Il lavoro di Metrangolo, Baldelli Bombelli, Cellesi pubblicato sulla prestigiosa rivista Chemistry A European Journal (https://doi.org/10.1002/chem.202002078) propone una nuova classe di co-polimeri fluorurati a base di acido poli lattico-co-glicolico (PLGA), un polimero biocompatibile e biodegradabile approvato dal Food and Drug Administration (FDA). In particolare, nei laboratori SupraBioNano diretti dal Professore Pierangelo Metrangolo (www.suprabionano.eu) sono stati sintetizzati e caratterizzati due nuovi composti, F3-PLGA e F9-PLGA, contenenti rispettivamente 3 e 9 atomi equivalenti di fluoro per consentire analisi di immagine molecolare mediante risonanza magnetica con fluoro 19 (19F-MRI). In Figura sono rappresentati i risultati ottenuti. Nel lavoro è stato dimostrato che i composti F-PLGA quando in soluzione acquosa formano spontaneamente nanoparticelle colloidali stabili caratterizzate da un forte segnale 19F-NMR. Inoltre, rispetto a nanoparticelle a base di PLGA non fluorurate, le nanoparticelle fluorurate hanno mostrato una maggiore capacità di incapsulare farmaci idrofobici contenenti atomi di fluoro quali il desametasone e la leflunomide. Studi preliminari in vitro su cellule umane hanno mostrato una significativa penetrazione cellulare e la capacità di rilasciare il farmaco incapsulato. Questo risultato, ottenuto grazie ai finanziamenti del progetto NEWMED, costituisce un importante passo per il raggiungimento dell’obiettivo 3 ‘Formulazione di nanovettori per il rilascio controllato di farmaci nel cuore malato’
